N6-甲基腺苷(m6A)是真核生物RNA分子上Z常见和Z丰富的修饰。m6A修饰由甲基转移酶复合体METTL3催化,并通过m6A RNA去甲基化酶FTO和ALKBH5去除,这些酶以α-酮戊二酸(α-KG)和Fe2+依赖的方式催化m6A去甲基化。已知METTL3、FTO和ALKBH5在许多生物过程中发挥重要作用,包括发育、代谢和生育。m6A占所有RNA碱基甲基化的80%以上,存在于不同物种中。m6A主要分布在mRNA中,也出现在非编码RNA中,如tRNA、rRNA和snRNA。m6A在mRNA转录本中的相对丰度已被证明可以影响RNA代谢过程,如剪接、核输出、翻译能力和稳定性以及RNA转录。由于m6A RNA甲基化酶和去甲基化酶缺陷引起的m6A甲基化水平异常可能导致RNA功能障碍并引起疾病。例如,由于FTO突变患者中FTO活性增加,通过尚未定义的途径,目标mRNA中m6A水平异常低,有助于肥胖及相关疾病的发生。RNA上动态可逆的化学m6A修饰也可能作为具有深远生物学意义的新表观遗传标记。因此,更多有助于更好理解m6A RNA甲基化水平和分布的有用信息,可以促进疾病的诊断和治疗。
目前,有几种方法用于表观转录组范围的m6A定位。这些方法包括MeRIP-seq、PA-m6A-seq、miCLIP和m6A-CLIP。MeRIP-seq已被广泛使用,但无法实现m6A分析的高分辨率。PA-m6A-seq、miCLIP和m6A-CLIP提高了分析分辨率,但存在可重复性差和过程复杂的问题。特别是,这些方法耗时(>2天)且成本高。
为了解决这些问题,EpigenTek开发了一种新方法:CUT&RUN RNA m6A-Seq(在目标下切割并使用核酸酶回收用于m6A测序)。我们的创新方法结合了MeRIP和m6A-CLIP的优势,具有z快的程序,并将其整合到EpiNext? CUT&RUN RNA m6A-Seq试剂盒中。
艾美捷 EpiNext? CUT&RUN RNA m6A-Seq试剂盒(P-9016)特点:
1、高富集度:使用RNA切割酶混合液同时切割RNA,并在目标m6A含有序列的两端切割/移除任何RNA序列,而不会影响抗体占据的RNA区域。仅与抗m6A抗体结合的短RNA片段被生成。因此,可以可靠地识别真正的目标m6A富集区域,并实现高分辨率定位。
2、低输入量:未结合的RNA切割和免疫捕获在同一个管中进行,这使得目标m6A含有区域得到Z大程度的保护,同时Z小化样本损失,允许输入RNA低至500纳克。
3、Z小背景:在目标m6A含有序列的两端切割未结合的RNA序列,能够Z小化MeRIP/测序背景,允许数据分析时读取数少于1000万次。
4、快速、简化的程序:从RNA到cDNA库的程序少于6小时,且操作时间少于1小时。
5、高度方便:试剂盒包含CUT&RUN RNA m6A-Seq的每个步骤所需的所有组分,这些组分足以进行m6A含有RNA序列捕获和捕获的cDNA库制备,因此使CUT&RUN RNA m6A-Seq成为Z方便、可靠且结果一致的方法。
CUT&RUN RNA m6A-Seq试剂盒检测原理:
EpiNext? CUT&RUN RNA m6A-Seq试剂盒包含了从总RNA开始进行成功的m6A-Seq所需的所有必要试剂。在反应中,目标m6A含有区域两端的RNA序列被切割/移除,并且使用结合有m6A捕获抗体的珠子捕获目标m6A含有片段。然后,目标m6A含有的RNA片段被回收、释放、逆转录,并连接接头。连接后的第一条链cDNA被扩增,然后用作文库合成的模板。使用高保真PCR混合液进行文库cDNA的扩增和索引。然后使用结合珠子清理cDNA。试剂盒中还包括一个阴性对照非免疫IgG,可以用来展示试剂盒的效果,并在富集RNA定量或生物分析仪分析步骤中展示性能。
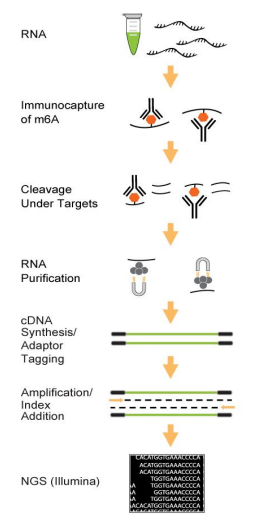
EpiNext?CUT&RUNRNA m6A-Seq试剂盒的工作流程。

微信扫码在线客服