CUT&RUN:
染色质免疫沉淀 (ChIP) 和 ChIP-seq 等能够比对蛋白-DNA 相互作用的方法的开发,使得人们越来越了解到异常的表观遗传调节可以引起许多人类疾病。核酸酶靶向切割和释放 (CUT&RUN) 是一项可用于染色质分析的新技术。
CUT&RUN 是一种使用靶标特异性一抗和Protein A - Protein G - 微球菌核酸酶 (pAG-MNase) 分离特异性蛋白-DNA 复合体的体内方法[1, 2, 3]。从细胞到 DNA 只需 1 至 2 天,并且它可以自动化,实现Z大通量和可重复性[4]。
为了分离目标蛋白-DNA 复合体,首先收集细胞,然后让其结合 Concanavalin A 包被的磁珠,以方便细胞处理,并在后续清洗过程中Z大程度减少细胞丢失。细胞膜用洋地黄皂苷透化,便于一抗进入胞核,在胞核,一抗会结合组蛋白、转录因子或目标辅因子。pAG-MNase 融合蛋白的 pAG 结构域随后结合一抗重链,从而使酶靶向至目标染色质区域。添加 Ca2+ 可激活 pAG-MNase,以启动 DNA 消化。这会使被切割的染色质复合体从基因组染色质中扩散出来、脱离胞核并进入样品上清液,使用 DNA 离心柱或苯酚/氯仿提取后再行乙醇沉淀的方法便可从上清液中收集这些被切割的染色质复合体。
纯化、富集的 DNA 随后使用 qPCR 检测和定量,或用下一代测序分析 (NGS) 与全基因组比对构建DNA 测序文库。
艾美捷CUT&RUN RNA m6A-Seq试剂盒(P-9016)检测原理:
EpiNext? CUT&RUN RNA m6A-Seq试剂盒包含了从总RNA开始进行成功的m6A-Seq所需的所有必要试剂。在反应中,目标m6A含有区域两端的RNA序列被切割/移除,并且使用结合有m6A捕获抗体的珠子捕获目标m6A含有片段。然后,目标m6A含有的RNA片段被回收、释放、逆转录,并连接接头。连接后的第一条链cDNA被扩增,然后用作文库合成的模板。使用高保真PCR混合液进行文库cDNA的扩增和索引。然后使用结合珠子清理cDNA。试剂盒中还包括一个阴性对照非免疫IgG,可以用来展示试剂盒的效果,并在富集RNA定量或生物分析仪分析步骤中展示性能。
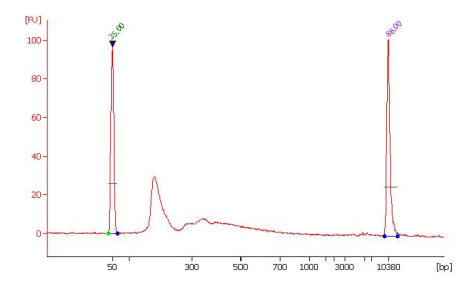
库片段的大小分布。用抗m6A抗体(EpigenTek,#A-1801)从3 ug总RNA中富集m6A片段,并用于DNA文库制备。195 bps的峰值反映了m6A抗体结合的RNA的插入大小(约55 bps)。
CUT&RUN相关文献:
1. Skene PJ, et al. Targeted in situ genome-wide profiling with high efficiency for low cell numbers. (2018) Nat. Protoc 13(5), 1006-1019. Pubmed29651053
2. Meers MP, et al. Improved CUT&RUN chromatin profiling and analysis tools.(2019) BioRxiv 1, 569129. bioRxiv 569129
3. Skene PJ and Henikoff S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. (2017) Elife 6, e21865. Pubmed28079019
4. Janssens DH, et al. (2018) Epigenetics Chromatin 22(1), 74. Pubmed30577869

微信扫码在线客服