【前情回顾】无需超声,Epigentek酶解法ChIP检测试剂盒(CUT&LUNCH)上新啦!
为什么还要花费数天来完成传统的CUT&RUN或ChIP分析?有了EpigenTek全新的CUT&LUNCH试剂盒,您只需一个上午2小时的时间,就能高效富集目标蛋白/DNA复合物,而无需在流程操作上费周章。 快速操作流程:在不到2小时内完成实验。 低非特异性背景:非特异性DNA结合仅为3-5%。 高特异性与高分辨率:在靶蛋白富集区域的两端选择性切割DNA。 成本效益高:与传统CUT&RUN相比,每次反应成本仅约为一半。 无缝整合现有流程:富集的DNA可直接用于NGS分析,可与现有且成熟的ChIP-seq生物信息学软件和工具无缝对接。 不再让冗长的实验步骤耽误您的工作进度。选择CUT&LUNCH,在满足时间和预算需求的前提下,获得高效且高质量的实验结果。让您的蛋白/DNA复合物富集更轻松省时,午餐前两小时就能搞定!
准备好加速您的染色质研究了吗? EpiNext CUT&LUNCH(Cleavage Under Target and Liberate Unique Nucleic Complex Homogenously)是一种新创的靶蛋白-DNA复合体定向切割与特异捕获技术,快速靶蛋白/DNA复合体富集 。基于该技术制备的试剂盒综合了ChIP和CUT&RUN的所有优势并克服其缺陷,可在最短时间内实现靶向蛋白/DNA复合物的富集。上午10点开始试验,12点完成,在午餐前就轻松搞定。
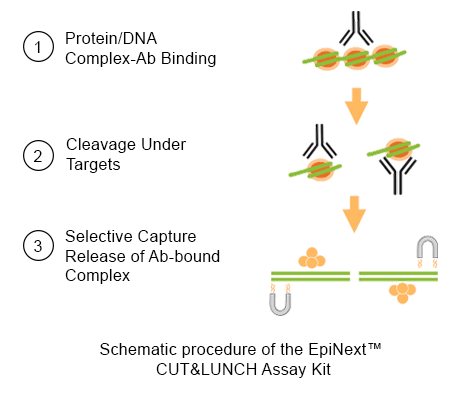
超快速三步法 超快速三步法示意图:
1. 蛋白-DNA复合体与抗体结合;
2. 靶向复合体定向酶切割;
3. 选择性捕获抗体结合的靶蛋白-DNA复合体 (add the images)
EpiNext CUT&LUNCH(Cleavage Under Target and Liberate Unique Nucleic Complex Homogeneously)分析试剂盒(货号P-2035)为研究体内DNA-蛋白相互作用提供了一种快速高效的方法。它整合了ChIP和CUT&RUN的所有优点,同时有效克服其局限性。
CUT&LUNCH分析具有如下优势与特点:
1 超快速三步法: 直接从完整细胞开始原位操作,无需ConA固定。整个流程仅需1小时50分钟,有效减少细胞核损伤与染色质丢失,同时保持天然染色质结构。
2 增强特异性与降低背景: 独特的核酸切割酶可在不影响蛋白结合DNA的前提下,于靶蛋白/DNA复合物两端选择性切割DNA,从而降低低序列偏好性和实现高特异性。非特异性蛋白-DNA复合物被有效清除,为目标蛋白富集区域的高分辨率定位提供保障。
3 低起始材料要求: 无论是游离DNA的高效切割还是免疫捕获步骤,都能在最短时间内完成。该方法既适用于细胞也适用于组织,并最大限度地保护目标蛋白不被降解,同时将样本损失降至最低。因此,起始细胞数可低至500个。
4 广泛适用性: 适用于多种培养细胞和新鲜或冷冻组织,并适用于多种物种。无论是组蛋白还是转录因子,都能在保持高特异性与灵敏度的前提下进行分析。
5 高度便捷: 试剂盒包含完成实验所需的全部组分,使EpiNext? CUT&LUNCH分析既方便又具成本效益。
6 无缝整合进现有分析流程: 富集的DNA可直接用于NGS测序,并可用现有、成熟的ChIP-seq生物信息学软件和工具进行分析。
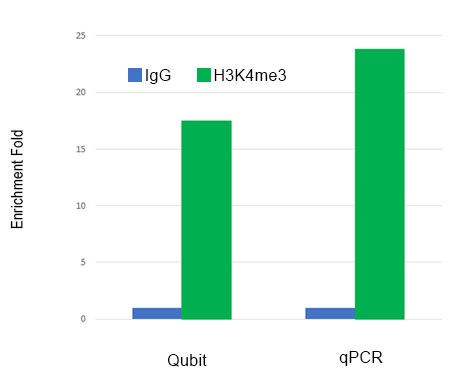 | 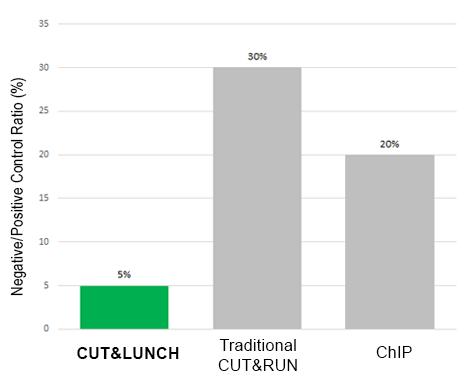 |
左图:使用EpiNext CUT&LUNCH试剂盒进行目标蛋白/DNA富集:通过阳性对照抗体(H3K4me3)从20万个MCF-7细胞中捕获组蛋白/DNA复合物。非免疫IgG作为阴性对照。富集的DNA被纯化并通过Qubit荧光定量或通过针对GAPDH启动子的qPCR分析进行富集倍数比较。右图:显著降低非特异性背景:CUT&LUNCH为5%,而CUT&RUN为30%,ChiP为20%。
CUT&LUNCH试剂盒(货号P-2035)包含完成从各种细胞样本中富集特定蛋白(组蛋白或高亲和力结合的转录因子)相关DNA复合物所需的全部试剂,通过qPCR或NGS对蛋白与DNA的相互作用进行分析。在本实验中,细胞首先被穿透化,并加入针对靶蛋白的高质量ChIP级抗体。然后利用独特的核酸切割酶混合物,对目标染色质区域两端的DNA进行特异性切割和去除。游离的非特异性蛋白-DNA复合物被清除,而结合有抗体的复合物将被选择性保留。最后,对捕获的蛋白/DNA复合物中的DNA片段进行纯化,可直接用于特定基因位点的qPCR分析或DNA文库构建,从而对蛋白与DNA的相互作用进行全基因组水平的精确解析。 起始材料 起始材料可为各种哺乳动物细胞,包括培养瓶或培养皿中的培养细胞、原代细胞、从血液或体液中分离的稀有细胞群、新鲜/冷冻组织中的细胞、以及从整体细胞群或胚胎细胞中分选得到的特定细胞类型等。每次反应的细胞数可在2 x 10^3至5 x 10^5之间。为获得最佳效果,推荐使用2 x 10^5个细胞作为起始量,但对修饰组蛋白的检测最低可至500个细胞。

CUT&LUNCH对比传统CUT&RUN和ChIP的优势
CUT&LUNCH | 传统CUT&RUN | ChIP | |
工作流程便利性 | 便利(仅19个步骤) | 较不便利(>50个步骤) | 不同ChIP实验时间和步骤变化较大 |
所需细胞数量 | 2,000 - 500,000 | 5,000 - 500,000 | 100 - 10,000,000 |
分析目标范围 | 适用于组蛋白和转录因子 | 适用于组蛋白,不适宜转录因子 | 适用于组蛋白和转录因子 |
已切割收集DNA的特异性 | 稳定,不受样本中目标分子数量影响 | 不稳定,受目标分子数量和类型影响 | 不适用 |
序列分辨率 | 高 | 高 | 中等 |
阴性对照与阳性对照比值(%) | 5% | 30% | 20% |
实验时间长度 | 1小时50分钟 | 6小时-2天 | 4小时-2天不等 |
单次实验反应价格 | 低 | 高 | 因实验类型不同而有差异 |
EpigenTek开发的改良型CUT&RUN技术——CUT&RUN-Fast,采用了一种新型独特的核酸切割酶混合物。该酶对序列偏好性极低,可同时对靶蛋白/DNA复合物两端的染色质进行切割和去除,并在不影响目标蛋白占位DNA的情况下,提升特异性和分辨率。这一改进大大加快了实验进程,无需过夜孵育。EpigenTek在CUT&RUN-Fast基础上进一步升级推出的CUT&LUNCH试剂盒,在极短的操作流程中实现了更高的特异性和更低的背景信号,让您可以在保持高特异性和高分辨率的前提下,大幅缩短实验时间并降低成本。

微信扫码在线客服