CUT&Tag试剂盒背景资料:
在体内富集组蛋白或转录因子(TF)复合的DNA,然后进行下一代测序,为研究全基因组蛋白质-DNA相互作用提供了有利的工具。它允许分析与活细胞中的DNA序列结合的特定蛋白质。这种分析要求该方法可靠地鉴定真正的靶蛋白富集区域,特别是从有限的细胞样品中。这些样品可以包括从组织中分离的稀有细胞群、从整个细胞群中分选的特定细胞和原代培养的亚群细胞如胚胎细胞。此外,该方法应确保富集的DNA包含Z小的背景,并且蛋白质-DNA结合区域的作图具有Z小的偏差和高分辨率。长期以来用于实现这一目标的主要方法是染色质免疫沉淀,然后测序(ChIP-Seq)。然而,ChIP的主要限制是它需要1)大量的输入材料,细胞或组织,以产生超过背景噪声的足够强的信号;和2)在初始固定步骤期间交联。有几种先进的方法可用于减少细胞数量或提高分辨率的ChIP-Seq。这些方法包括ChIP-exo、ChIPmentation。ChIP-exo提供了高分辨率的映射,但很耗时,并且需要大量的输入细胞。虽然ChIPmentation使用转座酶和测序兼容衔接子来实现ChIP过程中的连接整合,但它遵循传统的缓慢(2天)ChIP程序,无法实现高分辨率映射。
CUT&Tag试剂盒描述:
EpiNext? cTIP(CUT& Tag In-Place)测序试剂盒是 从哺乳动物细胞开始成功进行cTIP-Seq所需的一整套试剂。该试剂盒旨在从低输入细胞/染色质中富集蛋白质(组蛋白或强结合转录因子)特异性DNA复合物,并使用Illumina平台(如Illumina Genome Analyzer II、HiSeq和MiSeq系统)制备用于下一代测序的文库。创新的工作原理、优化的方案和试剂盒的组分允许以Z小化的非特异性背景水平捕获靶蛋白/DNA复合物,并快速构建非条形码(单重)和条形码(多重)文库,以便以较小的偏差和高分辨率映射靶蛋白-DNA相互作用区域。
艾美捷CUT&Tag试剂盒特点:
1、高浓缩: 使用独特的核酸切割酶混合物,其具有低序列偏倚,以同时片段化染色质并切割/去除靶蛋白/DNA复合物两端的任何DNA序列,而不影响靶蛋白占据的DNA。因此,可以可靠地鉴定真正的靶蛋白富集区域,并且可以实现高分辨率定位。
2、低投入材料: 稳健的无超声片段化、未结合DNA切割和免疫捕获均在同一单管中进行,并采用珠上连接。该方法允许在细胞和组织中使用,并允许以Z小的样品损失对靶蛋白进行Z大的降解保护。结果,输入细胞量可以少至500个细胞或染色质量可以低至50 ng。
3、原位标记: 在DNA纯化之前,DNA衔接子与染色质的珠上连接(原位)导致DNA片段大小增加,允许纯化与转录因子(TF)结合的非常短的片段(例如:< 70 bp)用于文库构建。
4、Z低背景: 在靶蛋白/DNA复合物的两(2)端原位切割未结合的DNA序列能够使免疫捕获/测序背景Z小化,从而允许使用<1000万个读数进行数据分析。
5、快速、简化的程序: 从细胞到文库DNA的过程不到5小时。
6、非常方便:该试剂盒包含CUT&Tag原位测序每个步骤所需的所有组件,足以用于蛋白质/DNA捕获和捕获的DNA文库制备,从而使cTIP(CUT&Tag In-Place)测序试剂盒Z方便,结果可靠且一致。
CUT&Tag试剂盒检测原理:
EpiNext? cTIP(CUT&Tag In-Place)测序试剂盒包含了从哺乳动物细胞开始进行成功的cTIP-Seq所需的所有必要试剂。在反应中,从细胞中分离出细胞核。目标蛋白-DNA复合物与感兴趣的ChIP级抗体结合/捕获。使用独特的核酸切割酶混合液,染色质被碎片化,目标蛋白/DNA复合物两端的DNA序列被切割/移除。在此过程中,被目标蛋白占据的DNA序列不受影响。接头被连接到珠子上与目标蛋白结合的DNA片段。然后,连接的DNA被释放、纯化,并使用高保真PCR混合液进行扩增,以构建文库DNA。DNA随后被清洁、释放和洗脱。试剂盒中包括一个阳性对照抗体(抗H3K9me3)、一个阴性对照非免疫IgG和对照染色质,这些可以用来在PCR/生物分析仪步骤中展示试剂盒的效果和性能。
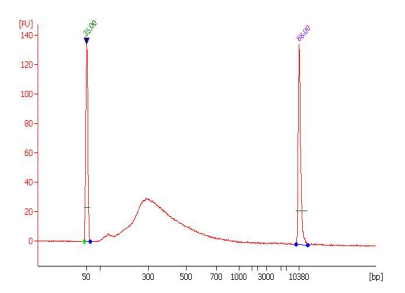
使用EpiNext?cTIP(CUT和Tag In Place)测序试剂盒对文库片段的大小分布:用对照抗体(H3K9me3)从Hela细胞分离的500 ng染色质中捕获组蛋白/DNA复合物,并用于DNA文库制备。295 bps的峰值反映了单核小体的插入大小(约150 bps)

微信扫码在线客服