Cleavage Under Target & Tagmentation (CUT&Tag)是一种新兴的DNA-蛋白质互作研究方法。CUT&Tag以融合了protein A/G的Tn5转座酶为核心,融合蛋白通过protein A/G与抗体结合,使得Tn5被栓系在靶位点周围,从而在目的位点附近进行酶切反应,并同时引入二代测序所必需的接头序列,产物DNA经过后续提取与PCR扩增后,得到直接用于测序的文库。CUT&Tag与传统的ChIP-Seq技术相比较,实验不需要超声打断,也不需要传统的连接法添加测序接头,操作简单、周期短、信噪比高、重复性好、细胞用量少,为极低起始细胞量和单细胞测序研究提供了可能性。
自2019年CUT&Tag技术诞生以来,经过大量的实践,发现该技术在组蛋白修饰及部分转录因子与DNA互作研究中可以获得高信噪比的结果,但是针对一些与DNA结合力较弱或者结合具有高度动态性的DNA结合蛋白,较难得到理想的实验结果。
艾美捷CUT&Tag试剂盒特点:
1、高浓缩: 使用独特的核酸切割酶混合物,其具有低序列偏倚,以同时片段化染色质并切割/去除靶蛋白/DNA复合物两端的任何DNA序列,而不影响靶蛋白占据的DNA。因此,可以可靠地鉴定真正的靶蛋白富集区域,并且可以实现高分辨率定位。
2、低投入材料: 稳健的无超声片段化、未结合DNA切割和免疫捕获均在同一单管中进行,并采用珠上连接。该方法允许在细胞和组织中使用,并允许以Z小的样品损失对靶蛋白进行Z大的降解保护。结果,输入细胞量可以少至500个细胞或染色质量可以低至50 ng。
3、原位标记: 在DNA纯化之前,DNA衔接子与染色质的珠上连接(原位)导致DNA片段大小增加,允许纯化与转录因子(TF)结合的非常短的片段(例如:< 70 bp)用于文库构建。
4、Z低背景: 在靶蛋白/DNA复合物的两(2)端原位切割未结合的DNA序列能够使免疫捕获/测序背景Z小化,从而允许使用<1000万个读数进行数据分析。
5、快速、简化的程序: 从细胞到文库DNA的过程不到5小时。
6、非常方便:该试剂盒包含CUT&Tag原位测序每个步骤所需的所有组件,足以用于蛋白质/DNA捕获和捕获的DNA文库制备,从而使cTIP(CUT&Tag In-Place)测序试剂盒Z方便,结果可靠且一致。
CUT&Tag试剂盒反应图示:
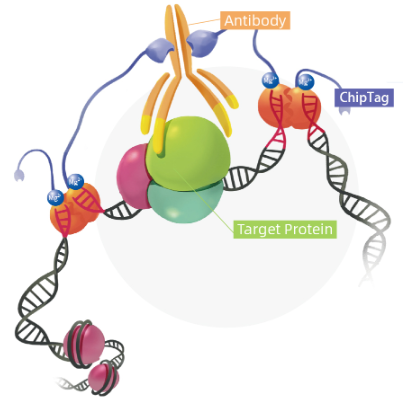
DNA-Target Protein-Antibody-ChipTag结合示意图

微信扫码在线客服